DBSCAN & クラスタリングの実用上の問題#
# 表形式のデータを操作するためのライブラリ
import pandas as pd
# 行列計算をおこなうためのライブラリ
import numpy as np
# 機械学習用ライブラリsklearnのKmeansクラス
from sklearn.cluster import KMeans
# 機械学習用ライブラリsklearnのDBSCANクラス
from sklearn.cluster import DBSCAN
# グラフ描画ライブラリ
import matplotlib.pyplot as plt
import matplotlib.colors as mcolors
import seaborn as sns;
sns.set(style='ticks')
%matplotlib inline
# ファイルの操作用
import os
# 警告文を表示させないおまじない
import warnings
warnings.filterwarnings('ignore')
クイズ#
Q1: クラスタ内誤差平方和#
第3章で用いた人工データ(University of Eastern Finlandの計算学部が公開しているデータセット)に対して,K-meansクラスタリングを適用することを考える. データを可視化すれば最適なクラスタ数は推測できるが,ここでは最適なクラスタ数は未知であると仮定する.
当該データをデータフレームに変換し,変数s1_dfに格納しなさい.
さらに,クラスタ数を3(K=3)としてs1_dfにK-meansクラスタリングを適用し,クラスタ内誤差平方和(SSE)を計算しなさい.
※ ヒント: scikit-learnライブラリを用いた場合,SSEの値はクラスタリング実行後,モデルのinertia_プロパティにアクセスすれば取得できる(参考).
url = "http://cs.joensuu.fi/sipu/datasets/s1.txt"
s1_df = pd.read_table(url, sep="\s+", header=None, names=['x', 'y'])
# 最初の10件のデータを表示
s1_df.head(10)
| x | y | |
|---|---|---|
| 0 | 664159 | 550946 |
| 1 | 665845 | 557965 |
| 2 | 597173 | 575538 |
| 3 | 618600 | 551446 |
| 4 | 635690 | 608046 |
| 5 | 588100 | 557588 |
| 6 | 582015 | 546191 |
| 7 | 604678 | 574577 |
| 8 | 572029 | 518313 |
| 9 | 604737 | 574591 |
K = 3
# クラスタリングを実行
model = KMeans(n_clusters=K, init='random')
model.fit(s1_df)
# SSEの計算
SSE = model.inertia_
SSE
213508776355610.8
Q2: エルボー法によるクラスタ数の決定#
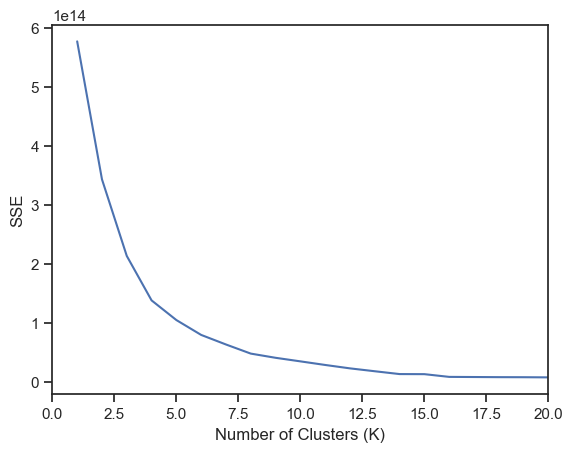
Q1におけるクラスタ数Kの値を1から20まで1ずつ変化させ,それぞれのKにおけるクラスタ内誤差平方和を求めなさい.
# SSEを格納するための変数
SSEs = []
for K in range(1, 21):
# クラスタリングを実行
model = KMeans(n_clusters=K, init='random')
model.fit(s1_df)
# SSEの計算
SSE = model.inertia_
print(f'K = {K} --> SSE = {SSE}')
# SSEを格納
SSEs.append(SSE)
K = 1 --> SSE = 576807041183705.2
K = 2 --> SSE = 343183593424387.25
K = 3 --> SSE = 213508903491353.16
K = 4 --> SSE = 138251485155489.56
K = 5 --> SSE = 104935513328366.2
K = 6 --> SSE = 79769282312091.64
K = 7 --> SSE = 63576882802374.34
K = 8 --> SSE = 48146924629521.44
K = 9 --> SSE = 41042047966160.41
K = 10 --> SSE = 35004560933478.71
K = 11 --> SSE = 28911271571376.125
K = 12 --> SSE = 23146652003743.785
K = 13 --> SSE = 18272499282576.816
K = 14 --> SSE = 13486733767168.47
K = 15 --> SSE = 13339552719882.875
K = 16 --> SSE = 8727376544840.372
K = 17 --> SSE = 8495356626590.272
K = 18 --> SSE = 8261106460001.316
K = 19 --> SSE = 8159804257250.533
K = 20 --> SSE = 7900878544599.463
Q3: エルボー法によるクラスタ数の決定#
Q2で計算したクラスタ内誤差平方和の値とエルボー法を用いて,Q1のデータにK-meansを適用する際の最適なクラスタ数を決めなさい.
ax = sns.lineplot(x=[K for K in range(1, 21)], y=SSEs)
ax.set_xlim((0, 20))
ax.set_xlabel("Number of Clusters (K)")
ax.set_ylabel("SSE")
plt.show()
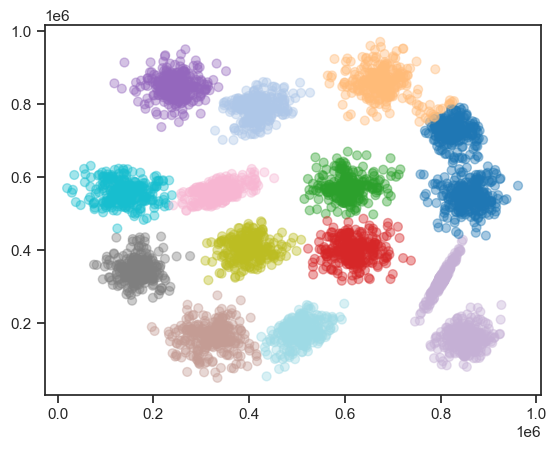
# クラスタリングを実行(K=13とした場合)
model = KMeans(n_clusters=13, init='random')
model.fit(s1_df)
# 結果を格納
labels = model.labels_
plt.scatter(
s1_df.x, s1_df.y,
c=labels,
alpha=0.4, # 透明度
s=40, # マーカーのサイズ
cmap="tab20_r" # カラーパレット(20色対応)
)
<matplotlib.collections.PathCollection at 0x33e900910>
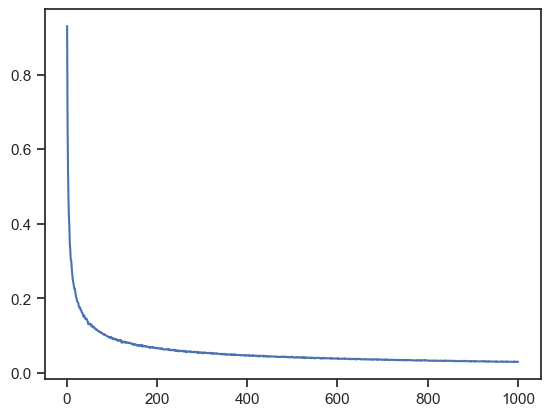
おまけクイズ#
Q4: データ間の距離#
以下の関数generateは,各次元の定義域が\([-1, 1]\)であるN次元空間に$K個のデータをランダムに生成する関数である.
generate関数はK行N列の行列を返す.
def generate(N, K):
return np.random.uniform(-1, 1, (K, N))
例えば,\(N=2\),\(K=10\)の場合,以下のようにgenerate関数は2次元のデータを10個生成し,行列形式で返す.
各行がデータ点に対応する.
generate(N=2, K=10)
array([[ 0.71922012, 0.79404868],
[ 0.8542639 , -0.38525764],
[-0.76125357, 0.31773887],
[ 0.75518281, -0.84001539],
[ 0.68740922, -0.44412347],
[-0.92083514, -0.66183711],
[ 0.39883398, 0.28542706],
[-0.97272434, -0.67785437],
[ 0.29322609, 0.82777789],
[-0.12805932, 0.40164165]])
generate関数を使ってランダムに生成された2次元の10個のデータにについて,全データ間のユークリッド距離を計算し,その結果を変数distに格納しなさい.
※ ヒント: scipyライブラリのspatial.distance.pdist関数を用いるとよい(参考).
# 点同士の距離を計算する関数をインポート
from scipy.spatial import distance
# データ生成
D = generate(N=2, K=10)
# 距離計算
dists = distance.pdist(D)
# 表示
dists
array([1.05965184, 0.52211314, 0.30829815, 1.07456711, 0.78142813,
1.02831203, 0.94178495, 0.68553864, 1.603236 , 1.51994411,
1.0063873 , 0.42035117, 0.28446327, 0.85969957, 0.92833385,
1.61625922, 1.9740061 , 0.52841058, 1.42806842, 1.25946985,
1.20991988, 1.08377411, 0.20084444, 1.3490191 , 0.90281022,
0.76454337, 0.75450816, 0.6527959 , 0.61005992, 1.32177749,
0.51140827, 0.47332886, 0.57326158, 1.47417951, 1.60869184,
0.82822971, 0.85507375, 1.37130382, 1.86909119, 0.13834446,
1.19504786, 1.13556505, 1.06089428, 1.04665613, 1.1767042 ])
Q5: データ間の距離の最小値と最大値#
Q4で求めた距離の最小値と最大値を求め,その差の絶対値max_dist_deltaを計算しなさい.
各次元の定義域がであるN次元空間においては,max_dist_deltaの最大値は\(2\sqrt{N}\)となる.
このことを踏まえて,最大値が1となるようmax_dist_deltaを正規化したnormalized_max_dist_deltaも計算しなさい.
※ ヒント: リストやnumpy.darrayの要素の最小値,最大値を計算するにはmin,max関数が使える.
補足: 距離の差を\(2\sqrt{N}\)で正規化するのか?#
各次元の定義域が[-1, 1]であるN次元空間における点同士のユークリッド距離がとりうる最大値は,
1次元空間では距離の最大値は\(2=2\sqrt{1}\)
2次元空間では距離の最大値は\(2 \sqrt{1^2 + 1^2}=2\sqrt{2}\)
3次元空間では距離の最大値は\(2 \sqrt{1^2 + 1^2 + 1^2}=2\sqrt{3}\)
となる. この観察から分かるように,N次元空間における点同士の距離がとり得る最大値は\(2\sqrt{N}\)となり,次元数が増えるにつれてとりうる距離の最大値が大きくなる. そのため,単純に最大距離と最小距離の差の「絶対値」を調べても,相対的に差が小さくなっているのか分からない. そこで,距離を\(2\sqrt{N}\)で割ることで,最大距離と最小距離の差を正規化(最大値が1になるように変換)する.
max_dist = max(dists)
min_dist = min(dists)
max_dist_delta = max_dist - min_dist
max_dist_delta
1.8356616439063096
import math
normalized_max_dist_delta = max_dist_delta / (2 * math.sqrt(2))
normalized_max_dist_delta
0.6490043981850985
Q6: 関数化#
Q4とQ5で行ったことを抽象化すると,以下のように書ける:
各次元の定義域が\([-1, 1]\)である\(N\)次元空間上で\(K\)個のデータをランダムに生成
各データ間のユークリッド距離の最小値と最大値の差を計算
ステップ2で求めた差を\(2\sqrt{N}\)で正規化した値(
normalized_max_dist_delta)を計算
ステップ1からステップ3を任意の回数だけ繰り返し,得られたnormalized_max_dist_deltaの平均値を\(\bar{\delta_{dist}}\)と定義する.
\(\bar{\delta_{dist}}\)を求める関数calc_mean_normalized_max_dist_deltaを定義しなさい.
なお,この関数の引数はN,K,iter(ステップ1からステップ3まで繰り返す回数)の3つとする.
def calc_normalized_max_dist_delta(N=2, K=10):
D = generate(N=2, K=10)
dists = distance.pdist(D)
max_dist = max(dists)
min_dist = min(dists)
max_dist_delta = max_dist - min_dist
return max_dist_delta / (2 * math.sqrt(N))
def calc_mean_normalized_max_dist_delta(N=2, K=10, iter=50):
max_dist_deltas = []
for i in range(iter):
max_dist_delta = calc_normalized_max_dist_delta(N, K)
max_dist_deltas.append(max_dist_delta)
return np.mean(max_dist_deltas)
calc_mean_normalized_max_dist_delta(N=100, K=50, iter=50)
0.0911202043953244
Q7: 次元の呪い#
Q6で定義した\(\bar{\delta_{dist}}\)について,\(K=50\),\(iter=100\)と固定して\(N\)を1から1000まで1ずつ変化させたときにどのように変化するか観察しなさい.
N_max = 1000
iter = 100
K = 50
means = []
for N in range(1, N_max + 1):
mean_normalized_max_dist_delta = calc_mean_normalized_max_dist_delta(N, K, iter)
means.append(mean_normalized_max_dist_delta)
# 折れ線グラフ上に結果を可視化
ax = sns.lineplot(x=list(range(1, N_max + 1)), y=means)
plt.show()
次元数が大きくなると,最大距離と最小距離の差がゼロに近づいていくことが分かる. 今回の実験ではN次元空間にデータがランダム(一様)に散らばっていることを想定していたが,実際はデータの分布には何らかの偏りがある. その場合でも,高次元空間ではデータ間の類似度・距離の扱いが難しいことには変わりはない. 高次元データは特徴選択・次元圧縮などを行って,低次元データに変換して機械学習を行う必要がある.
